Várias estirpes já existentes do SARS-CoV-2, bem como outras variantes que futuramente possam surgir, têm o potencial para escapar à resposta de células T citotóxicas do sistema imunológico das pessoas que sofram uma infeção. A conclusão é de um novo estudo de modelagem publicado, em 10 de fevereiro, na PLOS Computational Biology.
A resposta das células T em humanos é codificada geneticamente por moléculas HLA – isso significa que indivíduos diferentes têm HLAs diferentes, programados para reconhecer agentes patogénicos invasores com base em diferentes partes, ou “epitopos” dos agentes patogénicos. Com milhares de moléculas HLA diferentes na população humana e milhares de epitopos possíveis em qualquer vírus, a avaliação experimental da resposta imune de cada alelo HLA humano para cada variante viral não é viável. No entanto, métodos computacionais podem facilitar essa tarefa.
No novo estudo, os investigadores do Instituto de Saúde Carlos III, Espanha, determinaram primeiro o conjunto completo de epitopos de uma estirpe de referência original do SARS-CoV-2 de Wuhan, China. A equipa descobriu 1.222 epitopos de SARS-CoV-2 associados aos principais subtipos de HLA, cobrindo cerca de 90% da população humana; pelo menos 9 em cada 10 pessoas podem lançar uma resposta de células T à infeção de COVID-19 com base nesses 1.222 epitopos.
Os investigadores analisaram computacionalmente se algum dos 118.000 diferentes SARS-CoV-2 isolados em todo o mundo, descritos em um conjunto de dados do National Center for Biotechnology Information (NCBI), tinha mutações nesses epitopos. Os investigadores verificaram que 47% dos epitopos tinham sido mutados em pelo menos um SARS-CoV-2 isolado. Em alguns casos, os SARS-CoV-2 isolados existentes tinham mutações em várias regiões de epitopos, mas as mutações cumulativas nunca afetaram mais de 15% dos epitopos para qualquer tipo de alelo HLA.
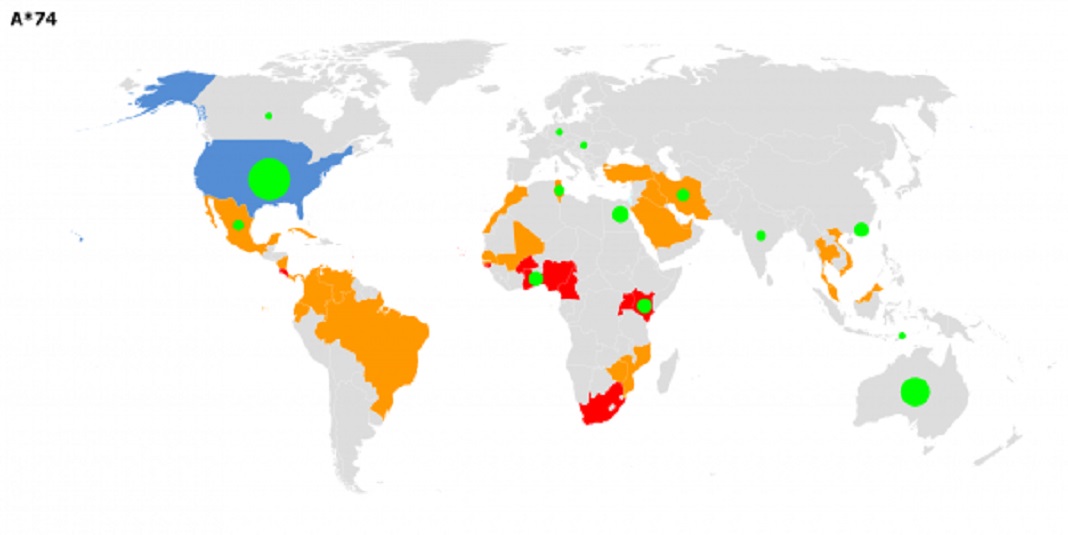
Quando a equipa de investigadores analisou alelos suscetíveis e a origem geográfica dos respetivos SARS-CoV-2, a equipa descobriu que eles coexistiam em algumas regiões geográficas – incluindo na África Subsaariana e no leste e sudeste da Ásia –, sugerindo uma potencial pressão genética sobre a resposta das células T citotóxicas nessas áreas.
“O acúmulo dessas mudanças em SARS-CoV-2 isolados independentes ainda é muito baixo para ameaçar a população humana global”, referem os investigadores, e acrescentam: “O nosso protocolo identificou mutações que podem ser relevantes para populações específicas e garantem uma vigilância mais profunda”.
No entanto, Antonio Martín-Galiano do Instituto de Saúde Carlos III, Espanha, observou que “mutações despercebidas do SARS-CoV-2” podem no futuro “ameaçar a resposta das células T citotóxicas em subpopulações humanas”.